Strategi kenalpasti & pengesahan Bingkai Bacaan Terbuka Pendek (sORF) dan Mikroprotein
-
Ditulis oleh: Profesor Madya Dr. Low Teck Yew
Tarikh Penerbitan: 23 Mei 2022
Pada Mac 2022, artikel ulasan kami yang bertajuk “Bingkai bacaan terbuka pendek (sORFs) dan mikroprotein: kemas kini tentang langkah pengenalan dan pengesahannya” (“Short open reading frames (sORFs) and microproteins: an update on their identification and validation measures”) (https://jbiomedsci.biomedcentral.com/articles/10.1186/s12929-022-00802-5) telah diterbitkan oleh Journal of Biomedical Science (https://jbiomedsci.biomedcentral.com/) [1]. Penerbitan ini adalah usaha kolektif Alyssa Leong, Dr. Lee Pey Yee, Dr. Aiman Mohtar, Dr. Saiful Effendi, dan Dr. Low Teck Yew dari UMBI. Dalam ringkasan ini, kami akan memberikan ringkasan tentang topik yang baru muncul ini.
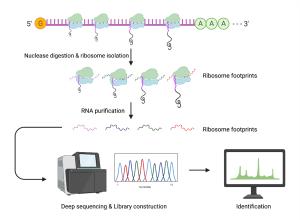
Bingkai bacaan terbuka pendek (sORFs) membentuk kurang daripada 300 bes, mengekod mikroprotein yang terdiri daripada kurang daripada 100 asid amino. Secara tradisinya, sORF ditolak oleh saluran paip anotasi genom sebagai bunyi yang tidak bermakna, dan sORF didapati bertaburan di pelbagai lokasi genom (Rajah 1). Kaedah utama di mana sORF boleh ditemui ialah pemprofilan ribosom (RIBO-Seq), yang merupakan satu pendekatan translatomik untuk mengukur aktiviti translasi selular dalam sel. RIBO-Seq berfungsi dengan mengasingkan segmen transkrip yang dilindungi oleh ribosom yang menterjemah secara aktif (Rajah 2). Melalui ini, sORF novel boleh dikesan dan diberi anotasi kepada kawasan genomik masing-masing. Berikutan penemuan yang menarik itu, banyak mikroprotein telah ditemui dan didapati terlibat dalam proses biologi penting, iaitu embriogenesis, pembezaan selular, dan tindak balas tekanan selular. Dengan kemajuan terkini dalam multi-omik, pengenalpastian, pengesahan dan pencirian fungsi sORF dan mikroprotein telah dapat dilaksanakan.
Rajah 1. Lokaliti sORF dalam genom dan transkrip
Rajah 2. Proses pemprofilan ribosom (RIBO-seq) di mana jejak ribosom diperoleh untuk penjujukan yang mendalam.
Sebaliknya, piawaian yang terbaik untuk pengenalpastian mikroprotein ialah pendekatan berasaskan spektrometri jisim (MS) (Rajah 3). Sebelum analisa dilakukan dengan spektrometri jisim, adalah penting untuk mengurangkan kerumitan sampel dengan pelbagai kaedah. Pertama, prinsip pengecualian saiz boleh digunakan untuk mengeluarkan protein bersaiz lebih besar, sekaligus memperkayakan protein bersaiz lebih kecil termasuk mikroprotein. Teknik yang biasa digunakan untuk pengecualian saiz ialah pemendakan asid, diikuti oleh penapis pemotongan berat molekul (MWCO), dan pengekstrakan fasa pepejal (SPE). Kedua, pengurangan kerumitan sampel boleh dicapai dengan pra-pemecahan sampel lisat protein menggunakan kromatografi interaksi tolakan-hidrofilik elektrostatik (ERLIC) dan pemfokusan isoelektrik resolusi tinggi (Hi-RIEF). Selepas pemerolehan MS, memberikan jujukan peptida dengan keyakinan tinggi adalah penting, dan ini bergantung pada liputan jujukan tinggi dan hingar latar belakang spektrum MS/MS yang rendah. Selain daripada pengenalpastian langsung dan kuantifikasi mikroprotein yang diterjemahkan, pendekatan berasaskan MS boleh menguraikan pengubahsuaian pasca terjemahan, yang menyimpulkan cerapan dalam fungsi biologi dan laluan isyarat.
Figure 3. Mass spectrometry-based approaches to isolate and identify microproteins.
Akhir sekali, pendekatan proteogenomik boleh mengenal pasti mikroprotein dengan menggabungkan data MS kepada data genomik, transkriptom dan/atau translatomik daripada sumber yang sama. Ini berguna untuk mengkaji genom yang belum diterokai atau separa berjujukan, seperti mikroprotein yang rendah dan kelas protein yang agak baharu dan tidak dicirikan. Selain itu, menggabungkan saluran paip pengiraan untuk menyokong data eksperimen apabila mencirikan peptida kecil beranotasi dan tidak bernotasi adalah penting. Terdapat beberapa pangkalan data yang tersedia secara terbuka khusus untuk sORF dan mikroprotein, seperti sORFs.org, SmProt dan OpenProt.
Berikutan pengenalpastian, pencirian fungsi mikroprotein boleh dilakukan dengan menggabungkan skrin CRISPR/Cas9 dan kajian interaksi protein-protein (PPI). Dengan memanipulasi ekspresi mikroprotein yang disasarkan, sejauh mana fenotip yang terhasil diubah menggambarkan betapa berpengaruhnya mikroprotein dalam laluan isyarat tertentu.
Manuskrip kami membincangkan bukan sahaja metodologi pengesanan, tetapi kami juga menyerlahkan cabaran dan penyelesaian yang berpotensi dalam mengenal pasti dan mengesahkan sORF dan mikroproteinnya. Kebaharuan ulasan ini terletak pada pengesahannya untuk peranan fungsi mikroprotein, yang boleh menyumbang ke arah landskap mikroproteomik masa depan.
Rujukan
- Leong AZX, Lee PY, Mohtar MA, et al (2022) Short open reading frames (sORFs) and microproteins: an update on their identification and validation measures. J Biomed Sci 29:. https://doi.org/10.1186/S12929-022-00802-5
-
Written by: Associate Professor Dr. Low Teck Yew
Published Date: 23 May 2022
In March 2022, our review article titled “Short open reading frames (sORFs) and microproteins: an update on their identification and validation measures” (https://jbiomedsci.biomedcentral.com/articles/10.1186/s12929-022-00802-5) was published by Journal of Biomedical Science (https://jbiomedsci.biomedcentral.com/) [1]. This publication is a collective effort of Alyssa Leong, Dr. Lee Pey Yee, Dr. Aiman Mohtar, Dr. Saiful Effendi, and Dr. Low Teck Yew from UMBI. In this post, we will provide a summary about this emerging topic.
Short open reading frames (sORFs) constitutes fewer than 300 bases, encoding microproteins comprising fewer than 100 amino acids. Traditionally dismissed by genome annotation pipelines as meaningless noise, sORFs were found to be scattered at various genome locations (Figure 1). The main method by which this was uncovered was ribosome profiling (RIBO-Seq), a translatomic approach to measure the cellular translational activity within a cell. RIBO-Seq functions by isolating a segment of transcript protected by the actively translating ribosome (Figure 2). Through this, novel sORFs can be detected and annotated to their respective genomic regions. Following the exciting discovery, numerous microproteins have been discovered and found to be involved in essential biological processes, namely embryogenesis, cellular differentiation, and cellular stress responses. With recent advancements in multi-omics, the identification, validation, and functional characterisation of sORFs and microproteins have become feasible.
Figure 1. Localities of sORFs in the genome and transcripts.
Figure 2. Ribosome profiling process where ribosome footprints are obtained for deep sequencing.
On the other hand, the gold standard for microprotein identification is mass spectrometry-based approaches (Figure 3). Before mass spectrometry, it is crucial to reduce sample complexities by various methods. First, size exclusion principles can be applied to remove the proteins of larger size, thus enriching for proteins of smaller sizes including the microproteins. Commonly used techniques for size exclusion are acid precipitation, followed by molecular weight cut off (MWCO) filters, and solid phase extraction (SPE). Secondly, reduction of sample complexity can be achieved by pre-fractionation of protein lysate samples using electrostatic repulsion-hydrophilic interaction chromatography (ERLIC) and high resolution isoelectric focusing (Hi-RIEF). Post MS acquisition, assigning peptide sequences with high confidence is crucial, and this depends on the high sequence coverage and low background noise of MS/MS spectra. Apart from the direct identification and quantification of translated microproteins, MS-based approaches can decipher post-translational modifications, which infers insights in biological functions and signalling pathways.
Figure 3. Mass spectrometry-based approaches to isolate and identify microproteins.
Finally, the proteogenomics approach can identify microproteins by combining MS data to genomic, transcriptomic and/or translatomic data from the same source. This is useful when it comes to studying unexplored or partially sequenced genomes, such as the microproteins being low-abundant and a relatively new and uncharacterised class of proteins. Moreover, incorporating a computational pipeline to corroborate with experimental data when characterising annotated and unannotated small peptides is crucial. There are several publicly available databases specialised for sORFs and microproteins, such as sORFs.org, SmProt and OpenProt.
Following identification, functional characterisation of microproteins can be done by incorporating CRISPR/Cas9 screen and protein–protein interaction (PPI) studies. By manipulating the expression of a targeted microprotein, the extent to which the resulting phenotype is altered depicts how influential the microprotein is in a particular signalling pathway.
Our review discusses not only detection methodologies, but we also highlight on the challenges and potential solutions in identifying and validating sORFs and their microproteins. The novelty of this review lies within its validation for the functional role of microproteins, which could contribute towards the future landscape of microproteomics.
Reference
- Leong AZX, Lee PY, Mohtar MA, et al (2022) Short open reading frames (sORFs) and microproteins: an update on their identification and validation measures. J Biomed Sci 29:. https://doi.org/10.1186/S12929-022-00802-5